PCA在Spark2.0用法比较简单,只需要设置:
.setInputCol(“features”)//保证输入是特征值向量
.setOutputCol(“pcaFeatures”)//输出
.setK(3)//主成分个数
注意:PCA前一定要对特征向量进行规范化(标准化)!!!
//Spark 2.0 PCA主成分分析
//注意:PCA降维前必须对原始数据(特征向量)进行标准化处理
package my.spark.ml.practiceimport org.apache.spark.ml.feature.PCA
import org.apache.spark.ml.feature.PCAModel
import org.apache.spark.ml.feature.StandardScaler
import org.apache.spark.sql.Dataset
import org.apache.spark.sql.Row
import org.apache.spark.sql.SparkSessionpublic class myPCA {public static void main(String[] args) {SparkSession spark=SparkSession.builder().appName("myLR").master("local[4]").getOrCreate()Dataset rawDataFrame=spark.read().format("libsvm").load("/home/hadoop/spark/spark-2.0.0-bin-hadoop2.6" +"/data/mllib/sample_libsvm_data.txt")//首先对特征向量进行标准化Dataset scaledDataFrame=new StandardScaler().setInputCol("features").setOutputCol("scaledFeatures").setWithMean(false)//对于稀疏数据(如本次使用的数据),不要使用平均值.setWithStd(true).fit(rawDataFrame).transform(rawDataFrame)//PCA ModelPCAModel pcaModel=new PCA().setInputCol("scaledFeatures").setOutputCol("pcaFeatures").setK(3)//.fit(scaledDataFrame)//进行PCA降维pcaModel.transform(scaledDataFrame).select("label","pcaFeatures").show(100,false)}
}
如何选择k值?
//PCA ModelPCAModel pcaModel=new PCA().setInputCol("scaledFeatures").setOutputCol("pcaFeatures").setK(100)//.fit(scaledDataFrame);int i=1;for(double x:pcaModel.explainedVariance().toArray()){System.out.println(i+"\t"+x+" ");i++;}
输出100个降序的explainedVariance(和scikit-learn中PCA一样):
1 0.25934799275530857
2 0.12355355301486977
3 0.07447670060988294
4 0.0554545717486928
5 0.04207050513264405
6 0.03715986573644129
7 0.031350566055423544
8 0.027797304129489515
9 0.023825873477496748
10 0.02268054946233242
11 0.021320060154167115
12 0.019764029918116235
13 0.016789082901450734
14 0.015502412597350008
15 0.01378190652256973
16 0.013539546429755526
17 0.013283518226716669
18 0.01110412833334044
...
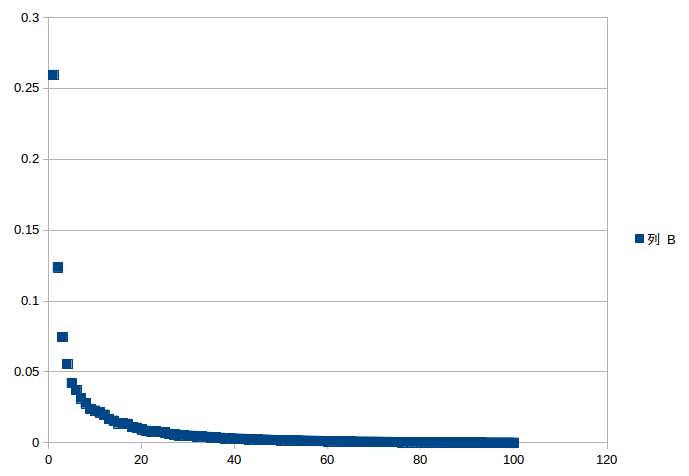
大约选择20个主成分就足够了
随便做一个图可以选择了(详细可参考Scikit-learn例子)
http://scikit-learn.org/stable/auto_examples/plot_digits_pipe.html
Scikit中使用PCA
参考http://blog.csdn.net/u012162613/article/details/42192293
sklearn.decomposition.PCA(n_components=None, copy=True, whiten=False)
参数说明:
n_components:
意义:PCA算法中所要保留的主成分个数n,也即保留下来的特征个数n
类型:int 或者 string,缺省时默认为None,所有成分被保留。
赋值为int,比如n_components=1,将把原始数据降到一个维度。
赋值为string,比如n_components=’mle’,将自动选取特征个数n,使得满足所要求的方差百分比。
copy:
类型:bool,True或者False,缺省时默认为True。
意义:表示是否在运行算法时,将原始训练数据复制一份。若为True,则运行PCA算法后,原始训练数据的值不 会有任何改变,因为是在原始数据的副本上进行运算;若为False,则运行PCA算法后,原始训练数据的 值会改,因为是在原始数据上进行降维计算。
whiten:
类型:bool,缺省时默认为False
意义:白化,使得每个特征具有相同的方差。关于“白化”,可参考:Ufldl教程
简单例子:
from sklearn import datasets
from sklearn.decomposition import PCAiris = datasets.load_iris()X = iris.data
y = iris.target
target_names = iris.target_namespca = PCA(n_components=3)
X_r = pca.fit(X).transform(X)
print "X_r"
print X_rprint "X"
print Xprint "pca.explained_variance_ratio"
print pca.explained_variance_ratio_